利用 Twist 寡核苷酸池在 RNA 疫苗开发中鉴定冠状病毒致病因子
今年,全球一直笼罩在 SARS-CoV-2 导致的 COVID-19 全球大流行带来的阴影之中。毫无疑问,它彻底改变了人们的生活。但经过我们不懈的努力,新的研究进展也给我们带来了希望。研究界受到的影响尤其显著,科学家们已经迅速动员起来努力寻找与该病毒感染、传播和变异等相关的答案。其中一个团队展示了如何利用 Twist 寡核苷酸池推动创新的方法,该方法有助于理解 SARS-CoV-2 如何入侵我们的免疫系统。
To cause infection, COVID-19 must enter a host cell and release its RNA genome. Virulence is established via replication of this genome, and production of new viral particles through the host cell’s expression of viral genes (Boopathi et al., 2020). Our cells have a number of innate mechanisms that restrict viral replication including RNA degrading proteins and genetic checkpoints alerting the immune system that “this cell has been infected.” These systems work in tandem with the adaptive immune system to halt virulence.
然而,在生存的军备竞赛中,某些病毒进化出了针对我们先天免疫系统的抗性。某些 RNA 病毒的基因组稳定性能保护其免受 RNA 降解酶的影响。RNA 通过自身碱基配对形成复杂的二级结构后,其稳定性有所增加。这些二级结构可能对宿主细胞的抗病毒机制不敏感,甚至可以主动阻断这种机制,从而使病毒不被察觉地复制。
靶向这种快速进化的病毒 RNA 的药物可以帮助宿主免疫系统有效降解并清除病毒感染。Wakida 等人最近在《Biochemical and Biophysical Research Communications》杂志上发表的一篇文章介绍了他们开发的一种方法,可以大量分析有利于病毒 RNA 在宿主细胞中的稳定性的相关序列。主要作者 Nobuyoshi Akimitsu 教授将这项研究描述为 RNA 药物向前迈进的重要一步。
“mRNA 不稳定,它们在细胞中的翻译往往很差。我们希望通过研究能够改善 mRNA 稳定性和结构的天然元件,增强 mRNA 作为药物的功能。”Akimitsu 教授解释道。
在这项研究中开发的方法称为“Fate-seq”。Fate-seq 的工作原理是稳定的 RNA 序列在哺乳动物细胞中的降解速度比不稳定序列慢得多。通过将目标 RNA 序列连接到信号分子(例如绿色荧光蛋白)并将其引入细胞,RNA 的降解可以被直接测量。一段时间后,荧光信号最强的细胞中的 RNA 结构最稳定。然后对信号最强的细胞进行测序,就可以精确定位高度稳定的 RNA 序列。
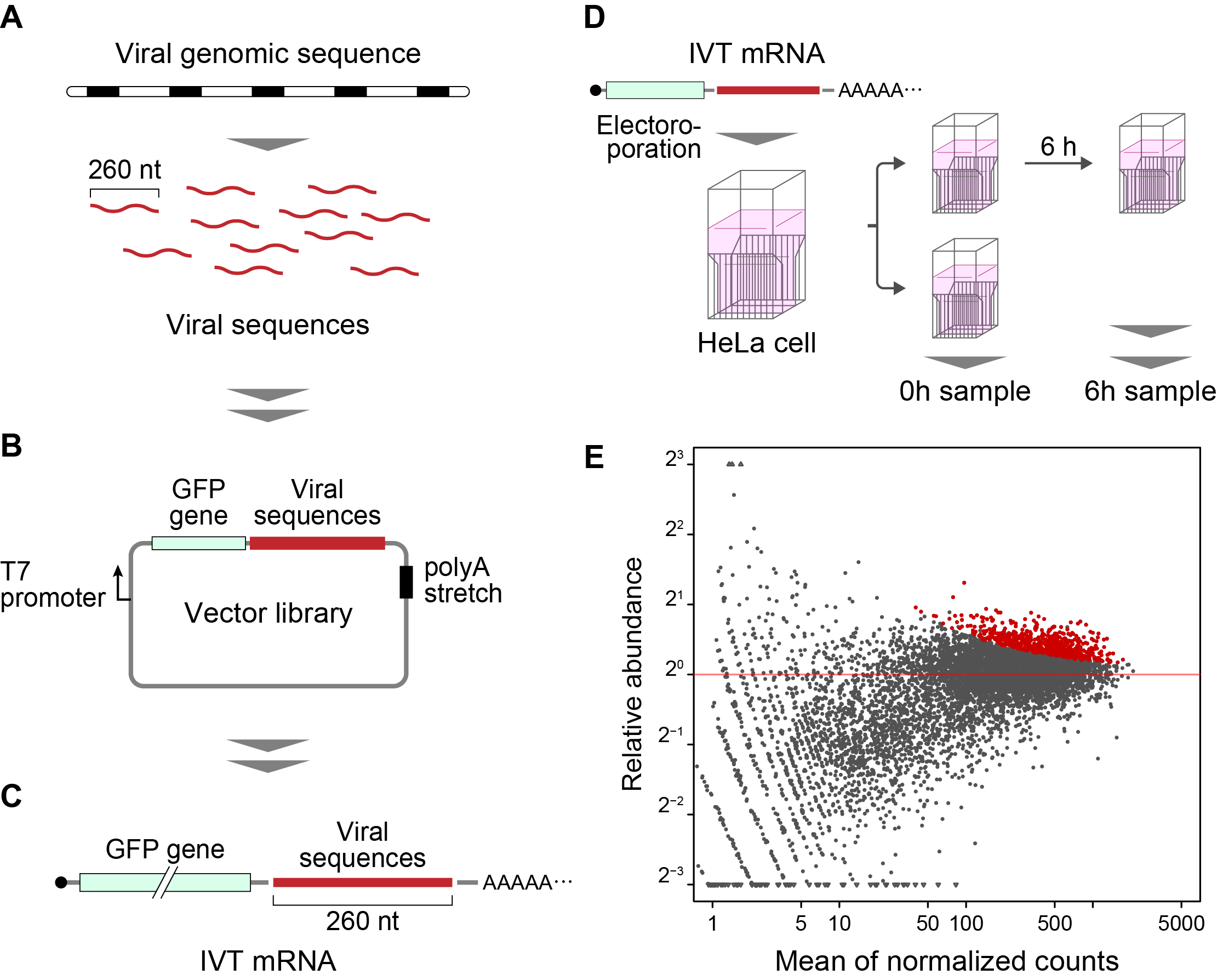
Wakida 等人使用 Twist Bioscience 寡核苷酸池检测了 26 种不同的病毒基因组。每个病毒基因组被分成了 260 个核苷酸 (nt) 的片段,其中包含 200 nt 的重叠。Akimitsu 教授说明了在该研究中使用高质量寡核苷酸的重要性。
“When doing a genome scan like Fate-seq, you need to be sure you are covering as much of the genome as possible, as evenly as possible. Because of the good uniformity and low error of Twist Oligo Pools, we can get even coverage across the viral genomes, and know we do not miss any potentially critical data.” Twist Oligo Pools can be synthesized up to 300 nucleotides, with an industry leading error rate of 1:2000.
在这项研究中,Fate-seq在严重急性呼吸综合征相关冠状病毒 (SARS-CoV) 和 SARS-CoV-2 中发现了两个高度保守的 RNA 稳定区域(COV001 和 COV002)。与 SARS-CoV 相比,Wakida 等人的结果确定了 SARS-CoV-2 中 COV001 二级结构的改变。作者的解读是“这种二级结构的差异可能对 SARS-CoV-2 的致病性有影响。这些鉴定出的序列可能会影响病毒 RNA 的稳定性以及与这些序列结合的 RNA 结合蛋白,它们也许能为治疗 COVID-19 等病毒感染性疾病提供潜在的靶点。”
Development of COVID-19 vaccines are currently in high-demand, many consisting of messenger RNA to activate the host immune system (Callaway, 2020). New technologies like Fate-seq to identify stabilizing molecules will offer significant benefits to the RNA vaccine development field, allowing more stable and efficacious vaccines to be produced. Moreover, Prof. Akimitsu reiterated “RNA stability is important for the viral life cycle. In theory, by inhibiting stability, we can reduce viral proliferation and virulence.” Fate-seq is therefore an expeditious and promising approach to spotlight critical genomic regions for further characterization as targets for antiviral therapeutics.
封面图片取自《Unsplash》的《Fusion Medical Animation》
您怎么认为?
喜欢
不喜欢
喜爱
惊讶
有兴趣
