生成 99% 所需的变异
使用单点基因突变文库的蛋白质工程筛选方法使研究人员可以探索方法蛋白质序列空间,研究序列与蛋白质结构和功能之间的关系。
Twist 定点饱和基因突变文库构建利用 Twist 专有硅基 DNA 合成平台进行大规模并行寡核苷酸合成。Twist 构建的文库经过 NGS 验证,确保所有目标突变呈现正确的突变率。
立即开始设计您的文库——专家团队随时准备帮助您设计完美的文库,以满足您的特定需求。点击此处订购和设计。
生成 99% 所需的变异
使用单点基因突变文库的蛋白质工程筛选方法使研究人员可以探索方法蛋白质序列空间,研究序列与蛋白质结构和功能之间的关系。
Twist 定点饱和基因突变文库构建利用 Twist 专有硅基 DNA 合成平台进行大规模并行寡核苷酸合成。Twist 构建的文库经过 NGS 验证,确保所有目标突变呈现正确的突变率。
深入了解 Twist 定点饱和基因突变文库 (SSVL) 如何通过探索序列空间来识别蛋白质结构和功能中的关键残基。
立即开始设计您的文库——专家团队随时准备帮助您设计完美的文库,以满足您的特定需求。点击此处订购和设计。
与传统方法(如简并法和 NNK 法)不同,定点饱和文库可消除密码子偏好性和不需要的替换。
TRIM 的重复产率很低,导致典型文库中全长产物 < 50%,而 Twist 文库产生更多可用变异,增加了有效文库大小。
| 易错的 PCR | 简并方法
(NNK/NNS) |
Twist 定点饱和 基因突变文库 |
|
|---|---|---|---|
| 消除序列偏倚 | 否 | 否 | 是 |
| 可用的密码子数量 | 未知 | 32 | 全部64 |
| 避免产生不必要的基序 | 否 | 否 | 是 |
| 可进行密码子优化 | 否 | 否 | 是 |
| 避免出现终止密码子 | 否 | 是 | 是 |
定点饱和基因突变文库可在筛选分析中对蛋白质的序列空间进行高效采样。
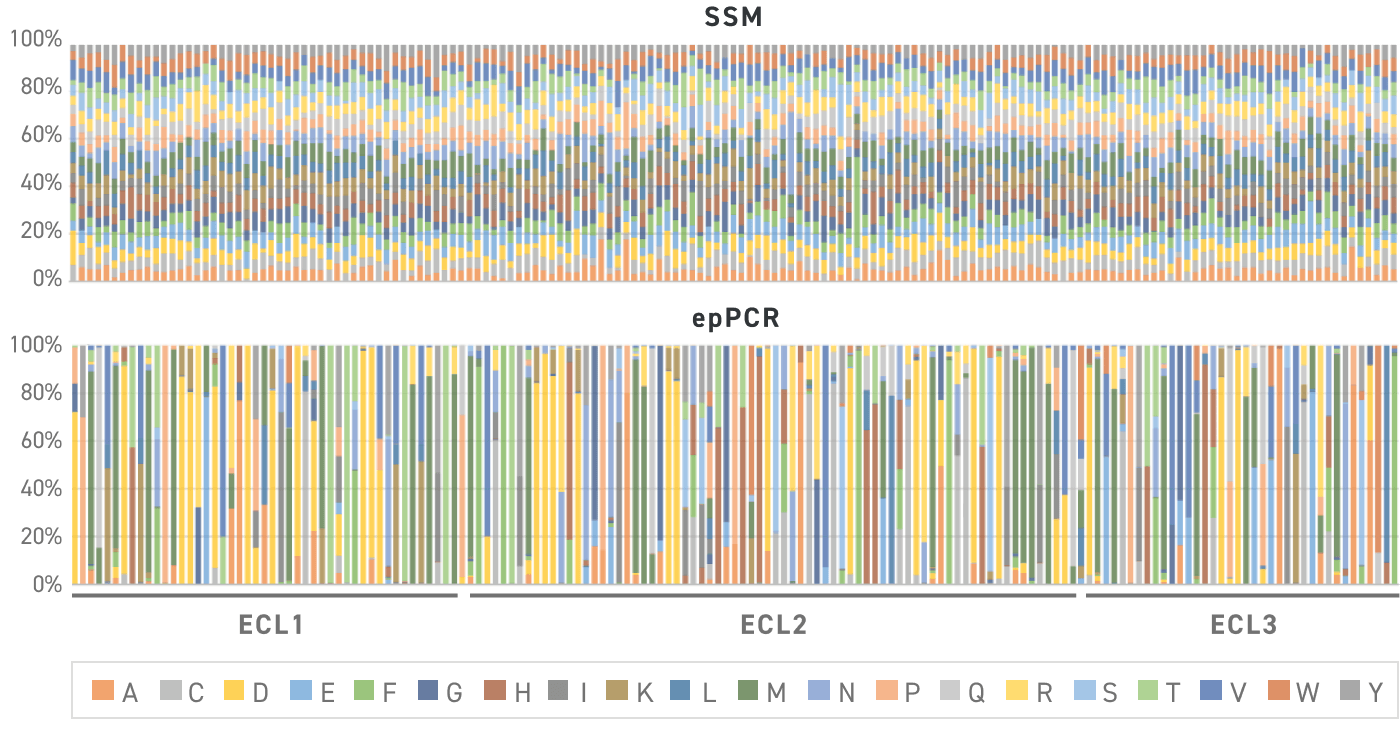
该图显示了分别使用 Twist SSVL 和易错的 PCR 构建时观察到的蛋白质中 65 个位置的氨基酸分布(每个位置 19 个预期变异)。条形代表不同的氨基酸位置,每种颜色表示观察到的变异频率。所有变异所占的比例都符合 Twist 文库预期。
与传统方法(如简并法和 NNK 法)不同,定点饱和文库可消除密码子偏好性和不需要的替换。
TRIM 的重复产率很低,导致典型文库中全长产物 < 50%,而 Twist 文库产生更多可用变异,增加了有效文库大小。
| 易错的 PCR | 简并方法
(NNK/NNS) |
Twist 定点饱和 基因突变文库 |
|
|---|---|---|---|
| 消除序列偏倚 | 否 | 否 | 是 |
| 可用的密码子数量 | 未知 | 32 | 全部64 |
| 避免产生不必要的基序 | 否 | 否 | 是 |
| 可进行密码子优化 | 否 | 否 | 是 |
| 避免出现终止密码子 | 否 | 是 | 是 |
定点饱和基因突变文库可在筛选分析中对蛋白质的序列空间进行高效采样。
该图显示了分别使用 Twist SSVL 和易错的 PCR 构建时观察到的蛋白质中 65 个位置的氨基酸分布(每个位置 19 个预期变异)。条形代表不同的氨基酸位置,每种颜色表示观察到的变异频率。所有变异所占的比例都符合 Twist 文库预期。

执行以下步骤
- 步骤 1:填写此页面上的联系方式表格
- 步骤 2:下载 SSVL 提交表并填写表格中的所有字段,以便进行您的文库设计
- 步骤 3:点击本页面的“提交文件”选项卡,上传完成的提交表格
准备好提交了吗?
- 步骤 1:上传完成的提交表格
- 步骤 2:我们的文库团队专家将审核您的项目
- 步骤 3:文库团队验证项目,并联系您提供报价
- 步骤 4:客户接受报价后,文库团队会将项目发送给制作团队
如果您有任何疑问,请随时发送电子邮件至 {{[email protected]}}
执行以下步骤
- 步骤 1:填写此页面上的联系方式表格
- 步骤 2:下载 SSVL 提交表并填写表格中的所有字段,以便进行您的文库设计
- 步骤 3:点击本页面的“提交文件”选项卡,上传完成的提交表格
准备好提交了吗?
- 步骤 1:上传完成的提交表格
- 步骤 2:我们的文库团队专家将审核您的项目
- 步骤 3:文库团队验证项目,并联系您提供报价
- 步骤 4:客户接受报价后,文库团队会将项目发送给制作团队
如果您有任何疑问,请随时发送电子邮件至 {{[email protected]}}
