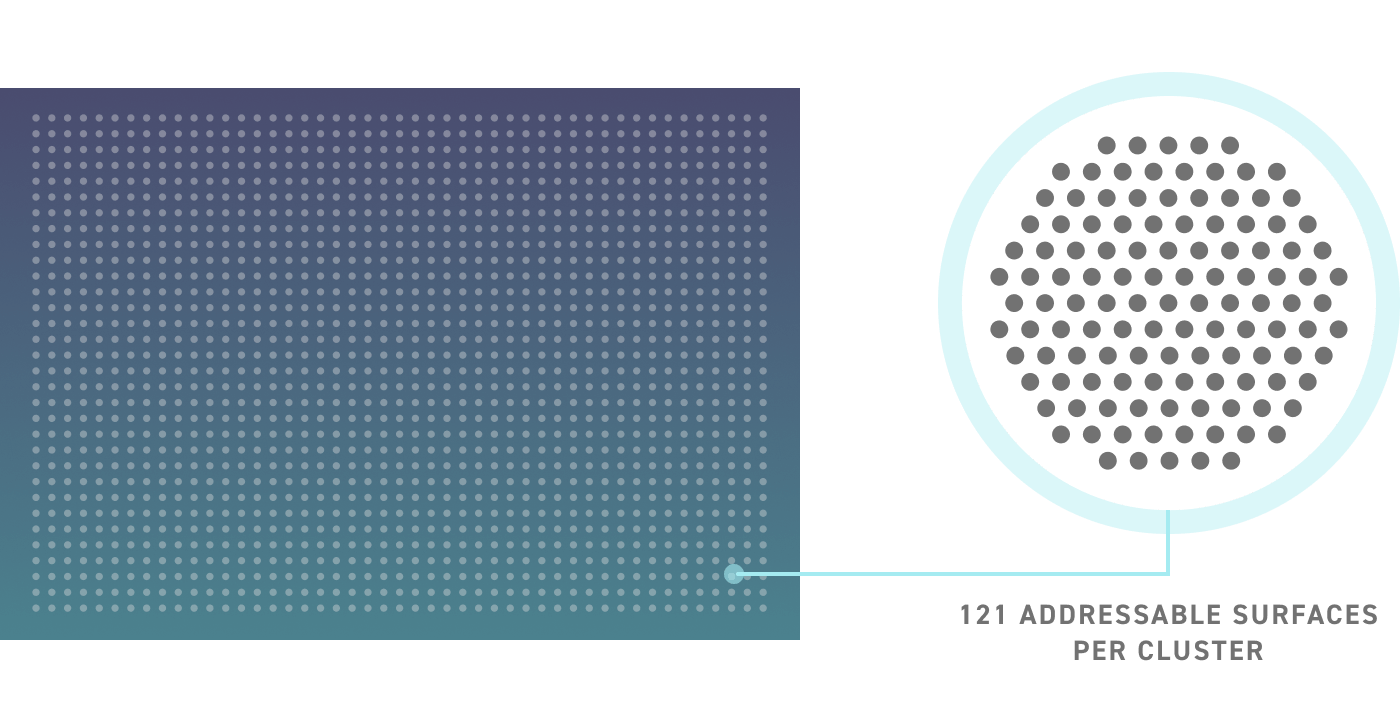
性能无与伦比,不惧任何规模
我们革命性的硅基 DNA 合成平台能够在单次运行中生成超过一百万个独特的 ssDNA 寡核苷酸。每个芯片包含数千个离散簇,每个簇包含 121 个可单独定位的表面,这些表面能够合成一个独特的寡核苷酸序列。硅芯片技术如下图所示。
由于芯片的 DNA 合成规模庞大,可以组装成池的独特寡核苷酸数量几乎没有限制。不要束缚于应用结果,订购为您的实验量身定制的寡核苷酸池。
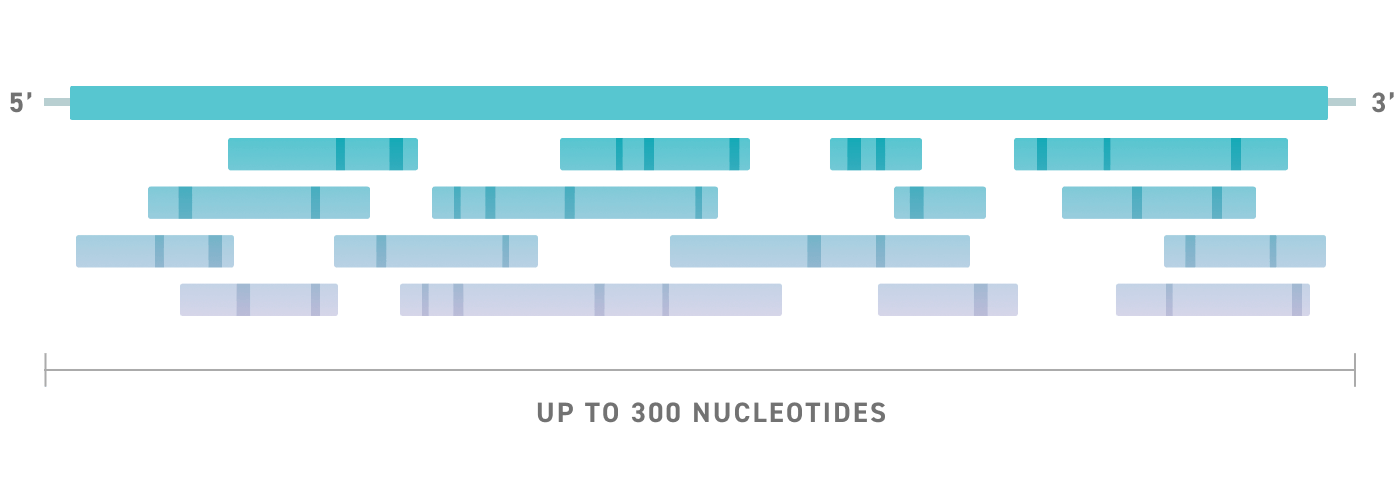
更多创新的空间
不为长度所束缚。对您所需的元件和序列进行编码,包括启动子、增强子、向导和探针在内统统囊括于扩展架构中。利用长达 300 nt 的寡核苷酸,可在单个设计中编码多种元件,方便您利用每个寡核苷酸实现更多需求。
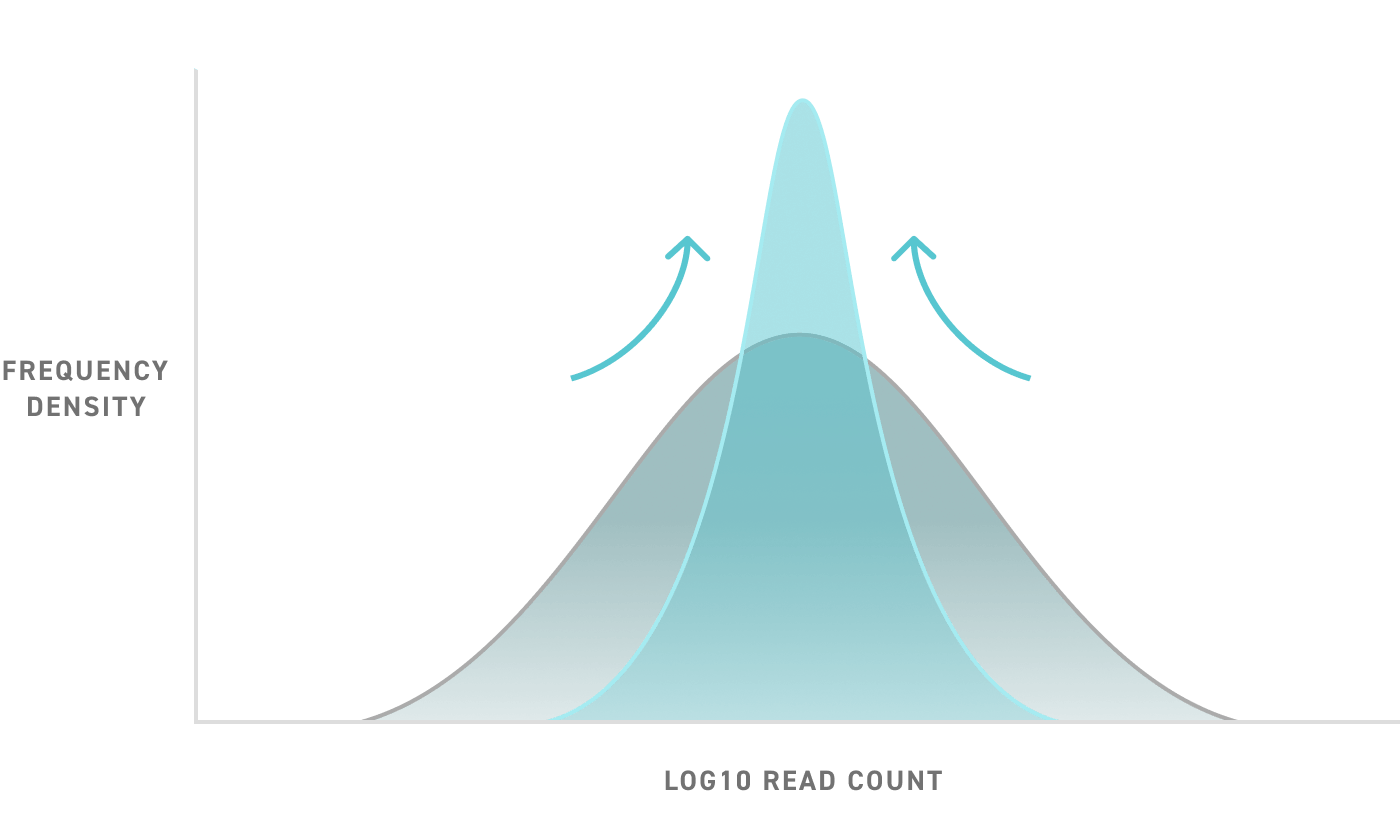
一次筛选,百分百正确
出色的寡核苷酸池均一度,无任何偏倚,每次筛选可提供更多特异性靶标。这意味着花费较少的精力就可达到您所需的数据覆盖度,节省时间和成本。
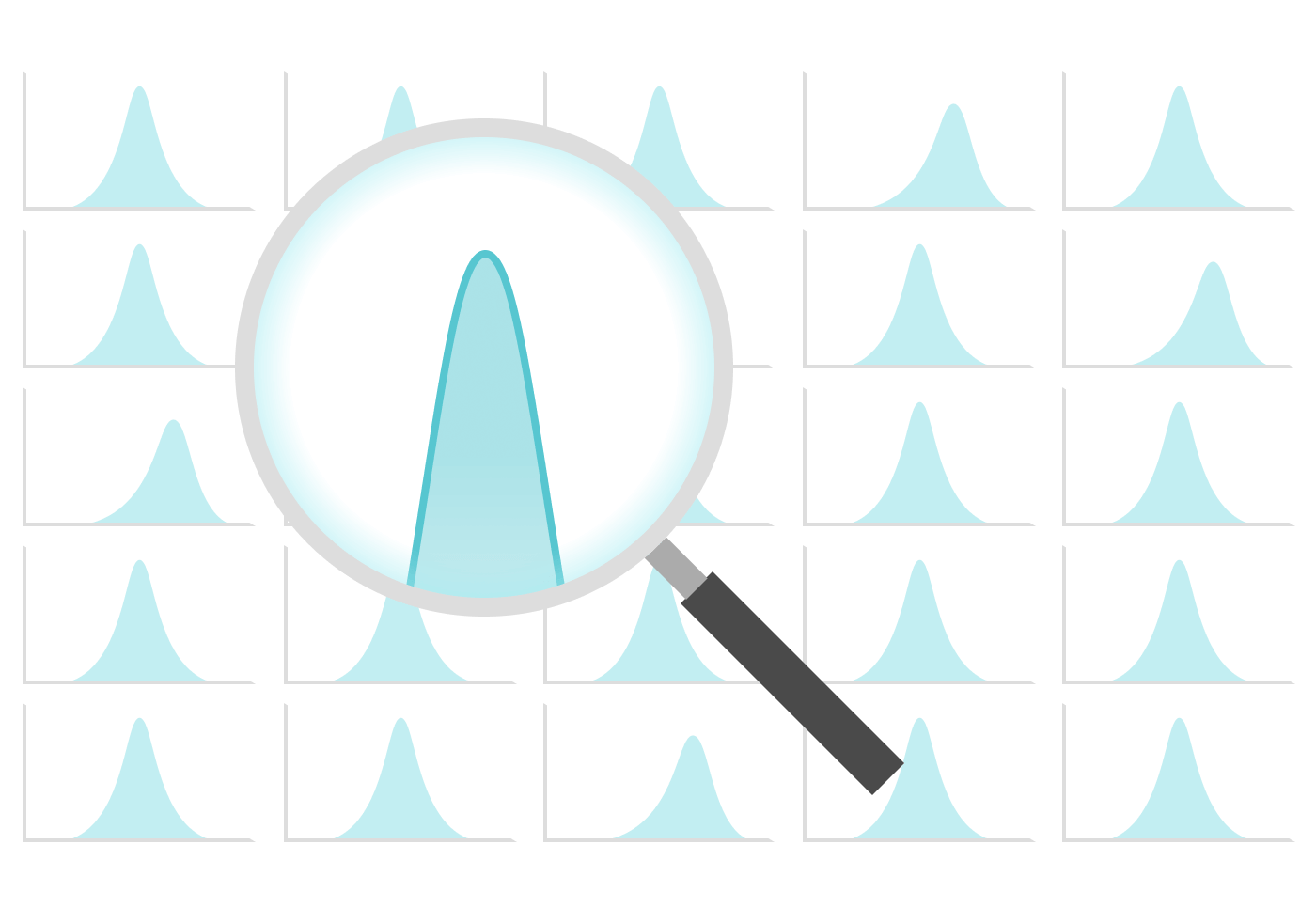
数据准,信心足
Twist 寡核苷酸池精准、均一,您可以充分信赖实验结果的质量。避免因为合成错误或效率低下而错过数据点。对于长度不超过 300 nt 的寡核苷酸,以及任何大小的池,都能实现行业领先的寡核苷酸占比和低错误率。您可根据应用所需的精准度和长度,来使用这些高品质的寡核苷酸池。
性能无与伦比,不惧任何规模
我们革命性的硅基 DNA 合成平台能够在单次运行中生成超过一百万个独特的 ssDNA 寡核苷酸。每个芯片包含数千个离散簇,每个簇包含 121 个可单独定位的表面,这些表面能够合成一个独特的寡核苷酸序列。硅芯片技术如下图所示。
由于芯片的 DNA 合成规模庞大,可以组装成池的独特寡核苷酸数量几乎没有限制。不要束缚于应用结果,订购为您的实验量身定制的寡核苷酸池。

更多创新的空间
不为长度所束缚。对您所需的元件和序列进行编码,包括启动子、增强子、向导和探针在内统统囊括于扩展架构中。利用长达 300 nt 的寡核苷酸,可在单个设计中编码多种元件,方便您利用每个寡核苷酸实现更多需求。
一次筛选,百分百正确
出色的寡核苷酸池均一度,无任何偏倚,每次筛选可提供更多特异性靶标。这意味着花费较少的精力就可达到您所需的数据覆盖度,节省时间和成本。

数据准,信心足
Twist 寡核苷酸池精准、均一,您可以充分信赖实验结果的质量。避免因为合成错误或效率低下而错过数据点。对于长度不超过 300 nt 的寡核苷酸,以及任何大小的池,都能实现行业领先的寡核苷酸占比和低错误率。您可根据应用所需的精准度和长度,来使用这些高品质的寡核苷酸池。

告诉我们您想详细了解哪些方面的信息。我们可以为您提供帮助!
联系我们
寡核苷酸池
Twist 寡核苷酸池是利用我们的硅基 DNA 编写技术合成的高度多样化的单链寡核苷酸集合。我们的合成平台能够在每次运行中大规模并行生产成千上万个高质量、高精度的寡核苷酸,从而生成复杂多样的寡核苷酸池,用于 CRISPR 筛选等应用。
工作原理
您向我们提供寡核苷酸序列,我们将合成您自主设计的寡核苷酸池文库,如此您能够将时间花费在实验和发现上。
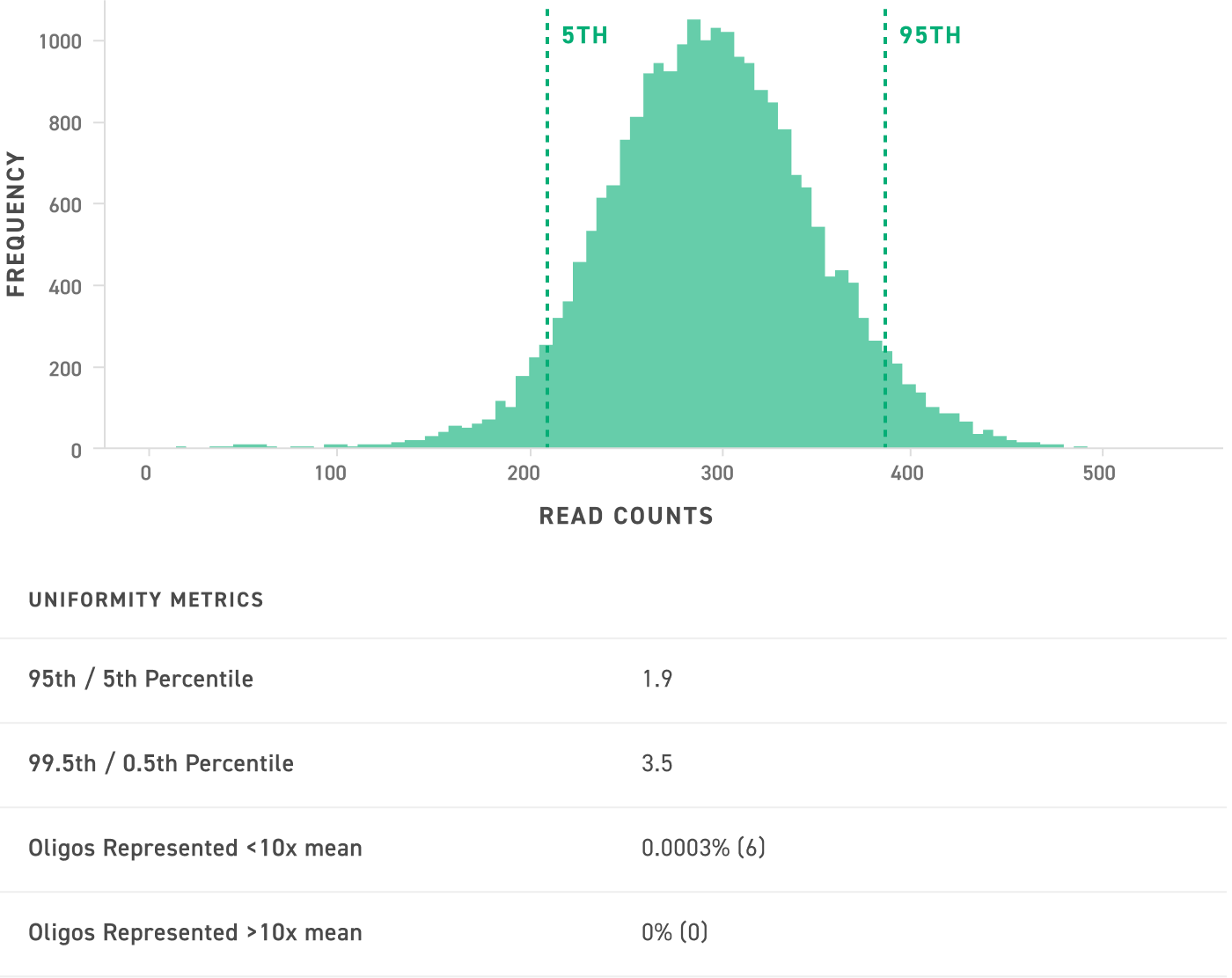
高度均一的寡核苷酸合成
来自典型寡核苷酸池(含 23,000 条 90 mer 寡核苷酸)的 NGS 质量控制数据展示了 300x 测序覆盖度下寡核苷酸池的均一性。对应表格表示该寡核苷酸池的均一性指标。Twist 寡核苷酸合成无偏倚,不仅具有高度均一性,而且能实现完全的寡核苷酸占比。
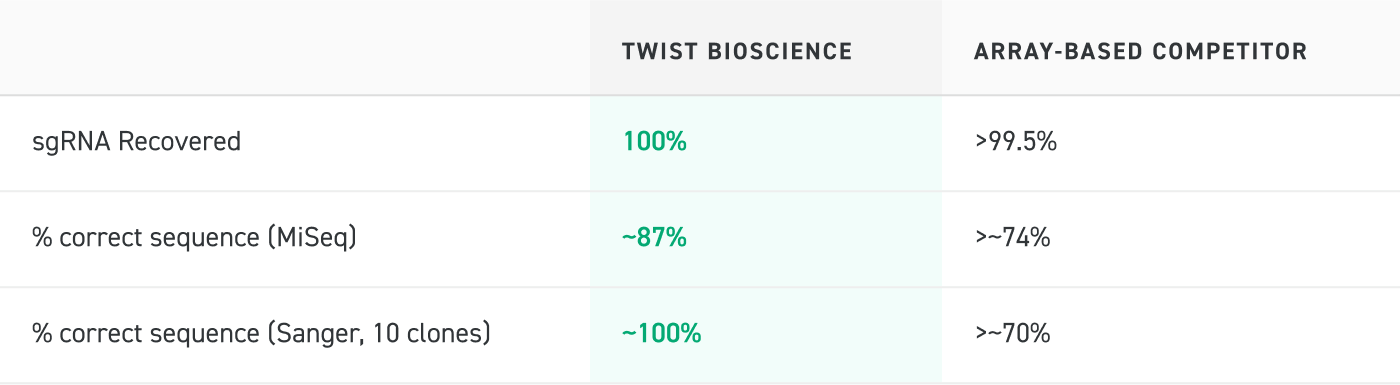
寡核苷酸池测序分析
对 Twist Bioscience 生产的寡核苷酸池和一种基于芯片的竞争产品进行测序分析,结果表明 Twist 寡核苷酸池包含 100% 的预期序列,而且正确序列百分比高于竞争产品。
规格
| 寡核苷酸长度 | 高达 300 nt |
| 寡核苷酸池大小 | 池的大小无限制 |
| 产量 | 每个寡核苷酸平均值 >0.2 fmol |
| 均一性 | 90% 以上的寡核苷酸在平均信号频数的 2.0 倍内出现 |
| 错误率 | 最多 1:3000 |
| 周转时间 | 仅需 3 天* |
| 价格 | 比同行优惠的定价 |
*标准寡核苷酸池产品的周转时间因长度和序列复杂性而异。寡核苷酸池在 20-121 个核苷酸长度的周转时间为 3-6 个工作日,在 121-200 个核苷酸长度的周转时间为 3-8 个工作日,及在 201-300 个核苷酸长度的周转时间为 5-10 个工作日。
告诉我们您想详细了解哪些方面的信息。我们可以为您提供帮助!
联系我们
寡核苷酸池
Twist 寡核苷酸池是利用我们的硅基 DNA 编写技术合成的高度多样化的单链寡核苷酸集合。我们的合成平台能够在每次运行中大规模并行生产成千上万个高质量、高精度的寡核苷酸,从而生成复杂多样的寡核苷酸池,用于 CRISPR 筛选等应用。
工作原理
您向我们提供寡核苷酸序列,我们将合成您自主设计的寡核苷酸池文库,如此您能够将时间花费在实验和发现上。

高度均一的寡核苷酸合成
来自典型寡核苷酸池(含 23,000 条 90 mer 寡核苷酸)的 NGS 质量控制数据展示了 300x 测序覆盖度下寡核苷酸池的均一性。对应表格表示该寡核苷酸池的均一性指标。Twist 寡核苷酸合成无偏倚,不仅具有高度均一性,而且能实现完全的寡核苷酸占比。

寡核苷酸池测序分析
对 Twist Bioscience 生产的寡核苷酸池和一种基于芯片的竞争产品进行测序分析,结果表明 Twist 寡核苷酸池包含 100% 的预期序列,而且正确序列百分比高于竞争产品。

规格
| 寡核苷酸长度 | 高达 300 nt |
| 寡核苷酸池大小 | 池的大小无限制 |
| 产量 | 每个寡核苷酸平均值 >0.2 fmol |
| 均一性 | 90% 以上的寡核苷酸在平均信号频数的 2.0 倍内出现 |
| 错误率 | 最多 1:3000 |
| 周转时间 | 仅需 3 天* |
| 价格 | 比同行优惠的定价 |
*标准寡核苷酸池产品的周转时间因长度和序列复杂性而异。寡核苷酸池在 20-121 个核苷酸长度的周转时间为 3-6 个工作日,在 121-200 个核苷酸长度的周转时间为 3-8 个工作日,及在 201-300 个核苷酸长度的周转时间为 5-10 个工作日。
告诉我们您想详细了解哪些方面的信息。我们可以为您提供帮助!
联系我们
新的克隆寡核苷酸池
寡核苷酸池的质量是实验成功的基础。合成或克隆过程中的错误很容易使寡核苷酸池的质量出现偏差,导致所需序列的表达过度和表达不足。Twist 现在提供优化的克隆服务,这使您可以进一步避免测试 PCR 扩增条件、选择正确的聚合酶和引物对以及设计克隆工作流程所带来的困难。避开繁琐的流程,让 Twist Bioscience 为您合成和克隆寡核苷酸池。
工作原理
获得您定制的克隆寡核苷酸池仅需两个步骤。您向我们提供寡核苷酸序列,我们将合成、扩增并克隆您自主设计的寡核苷酸池文库,如此您能够将更多时间集中在实验和发现上。
Twist Bioscience 不同长度的克隆寡核苷酸池均具有高均一性和低错误率
长度小于 150 个核苷酸(即使具有较高 GC 含量)扩增和克隆的寡核苷酸池的性能数据:
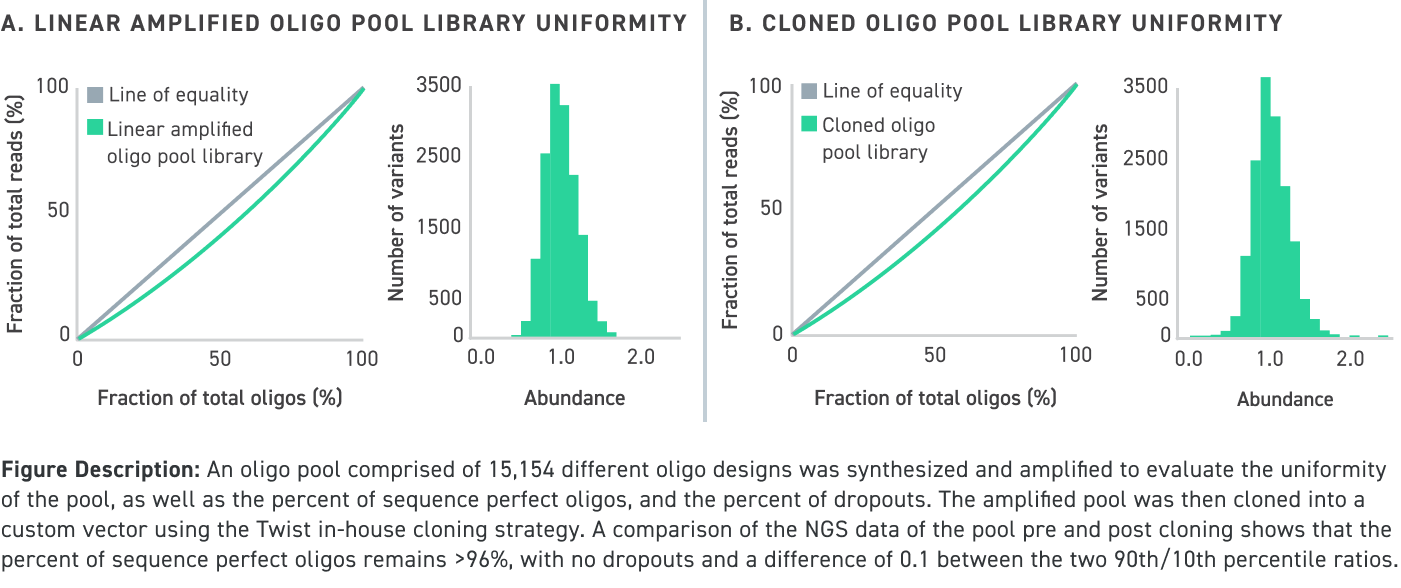
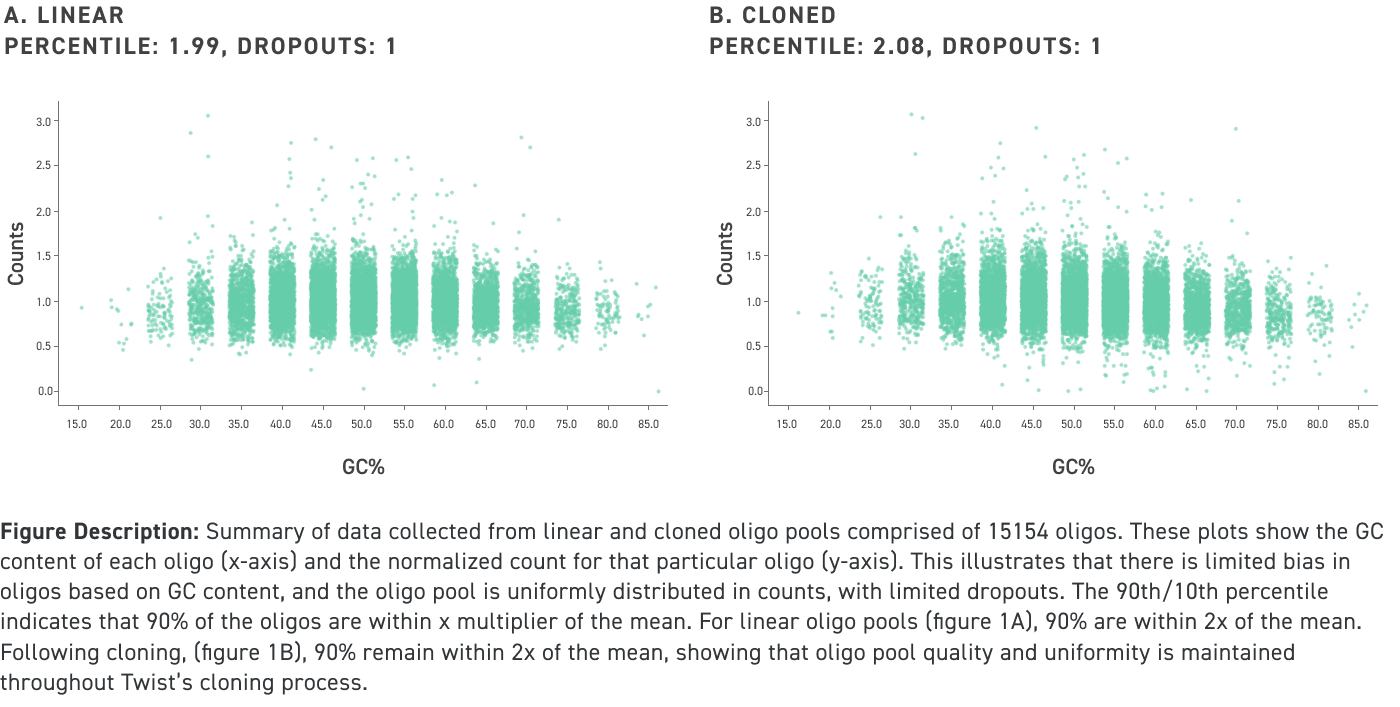
长达 300 个核苷酸(即使具有较高 GC 含量)扩增和克隆的寡核苷酸池的性能数据:
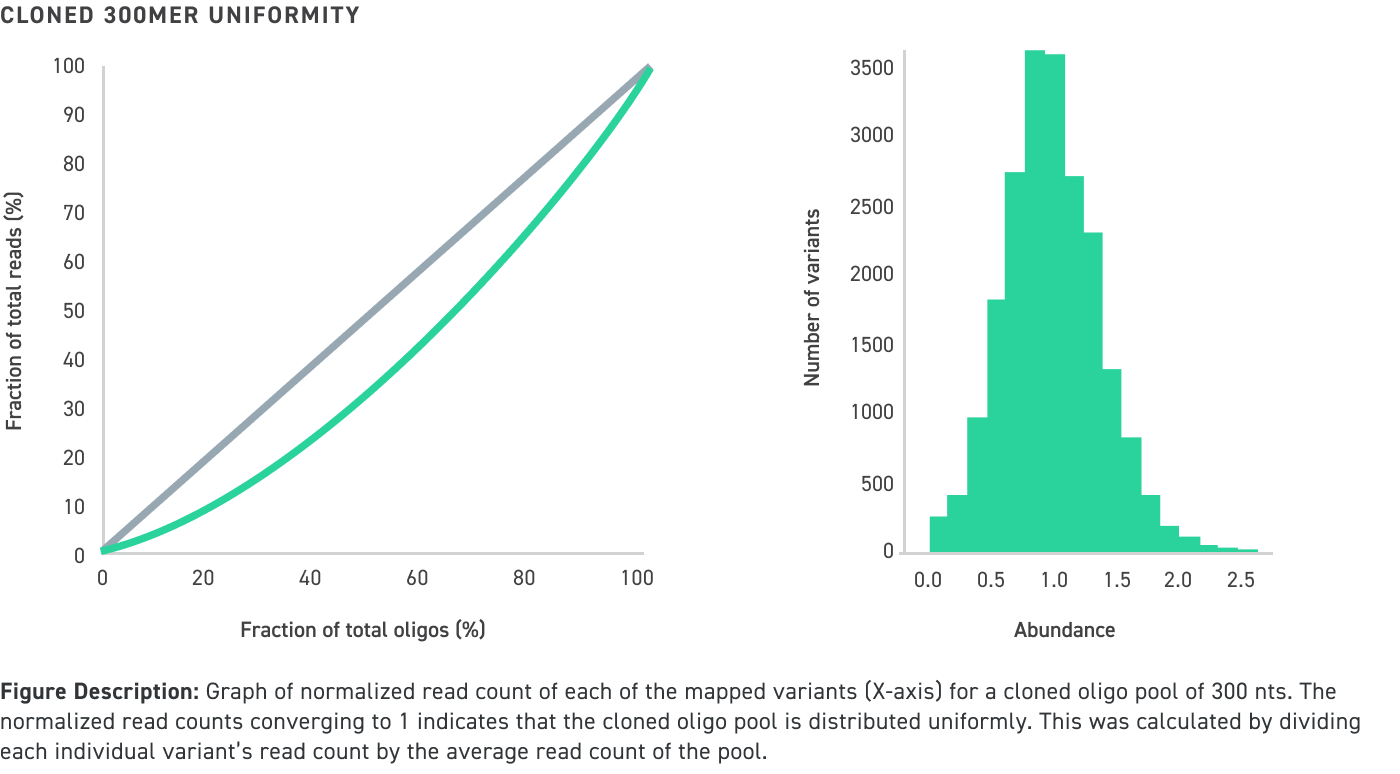
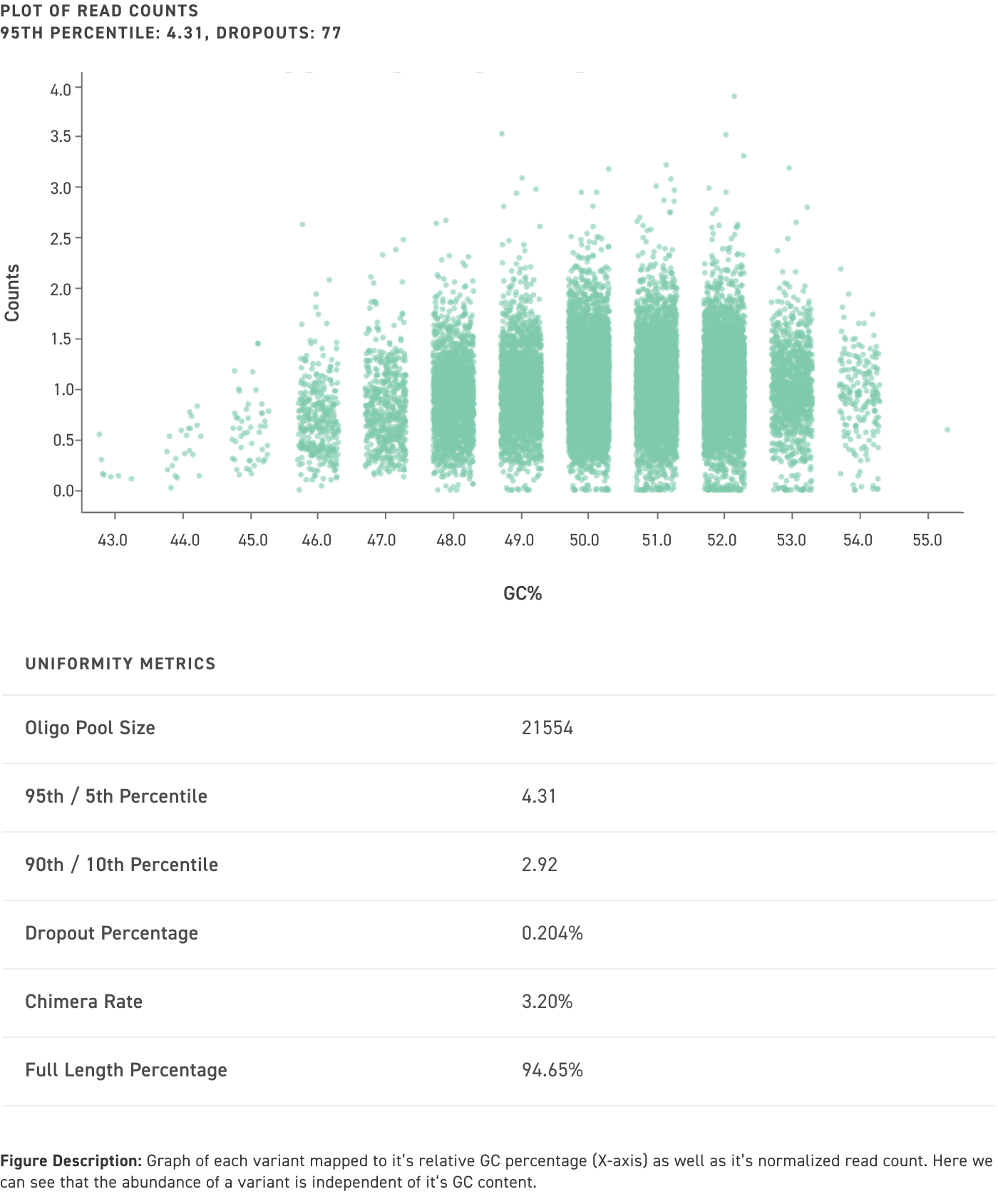
在实验数据中呈现出色的质量和准确性
Twist 的克隆寡核苷酸池不仅错误率低、均一性高,而且嵌合率也很低。嵌合体是不需要的杂交分子,是由于 PCR 条件不理想而导致寡核苷酸池中不同寡核苷酸的不当扩增和重组而产生的。请参阅下面的插图,了解中靶克隆池与包含嵌合体的脱靶克隆池(最终形成由不需要的序列组成的克隆寡核苷酸池)的示例:
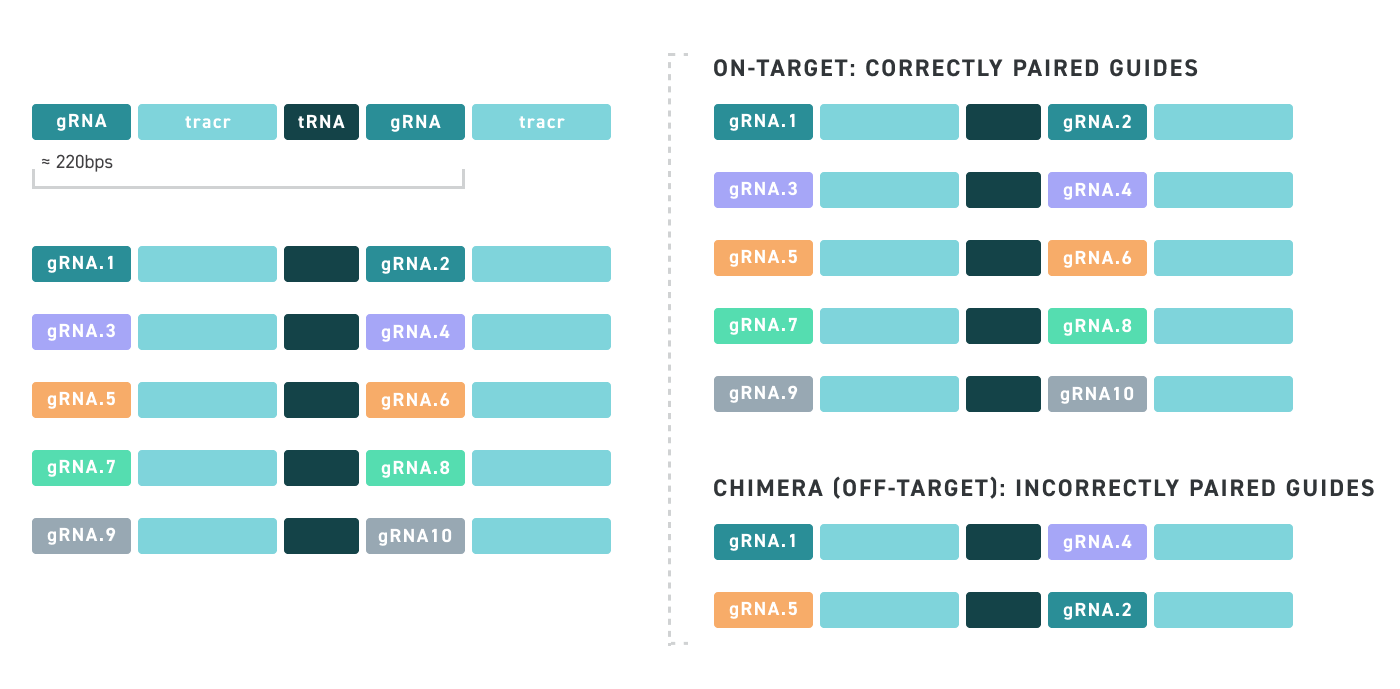
使用 Twist 产品,您再也不必担心嵌合了!查看以下示例,获得更直观的了解:
传统扩增与 Twist 内部扩增方法的性能对比
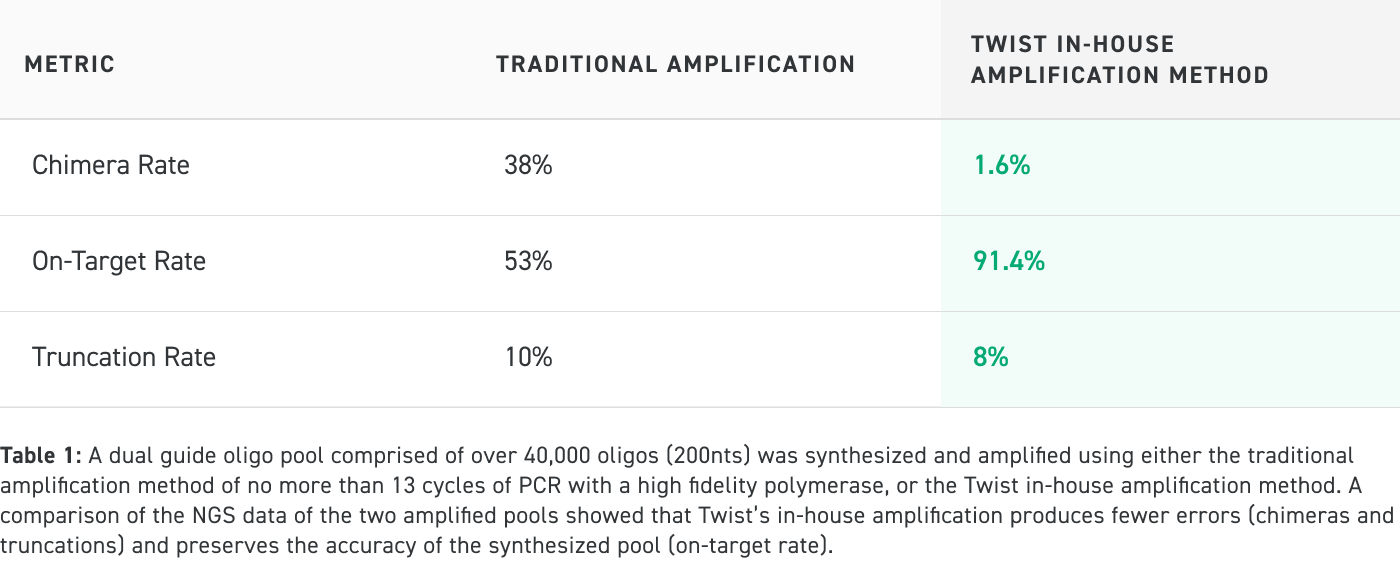
规格
| 寡核苷酸长度高达 300nt |
| 寡核苷酸池大小 池大小无限制 |
| 每个寡核苷酸平均产量 > 0.2 fmol |
| 均匀度 > 90% 寡核苷酸(在平均值的 < 5.0 倍范围内) |
| 嵌合率低至 1.5%* |
| 周转时间缩短至 4 周* |
*条款和条件:克隆寡核苷酸池(包括最多 100 个和 300 个核苷酸的克隆和扩增)的基线周转时间分别为 4-6 周和 6-8 周。对于所有需要新载体加入的克隆寡核苷酸池,无论长度如何,此基线周转时间将额外增加 2 周。嵌合率、丢失率和均一性将会根据序列复杂性而产生变化。
告诉我们您想详细了解哪些方面的信息。我们可以为您提供帮助!
联系我们
新的克隆寡核苷酸池
寡核苷酸池的质量是实验成功的基础。合成或克隆过程中的错误很容易使寡核苷酸池的质量出现偏差,导致所需序列的表达过度和表达不足。Twist 现在提供优化的克隆服务,这使您可以进一步避免测试 PCR 扩增条件、选择正确的聚合酶和引物对以及设计克隆工作流程所带来的困难。避开繁琐的流程,让 Twist Bioscience 为您合成和克隆寡核苷酸池。
工作原理
获得您定制的克隆寡核苷酸池仅需两个步骤。您向我们提供寡核苷酸序列,我们将合成、扩增并克隆您自主设计的寡核苷酸池文库,如此您能够将更多时间集中在实验和发现上。

Twist Bioscience 不同长度的克隆寡核苷酸池均具有高均一性和低错误率
长度小于 150 个核苷酸(即使具有较高 GC 含量)扩增和克隆的寡核苷酸池的性能数据:


长达 300 个核苷酸(即使具有较高 GC 含量)扩增和克隆的寡核苷酸池的性能数据:


在实验数据中呈现出色的质量和准确性
Twist 的克隆寡核苷酸池不仅错误率低、均一性高,而且嵌合率也很低。嵌合体是不需要的杂交分子,是由于 PCR 条件不理想而导致寡核苷酸池中不同寡核苷酸的不当扩增和重组而产生的。请参阅下面的插图,了解中靶克隆池与包含嵌合体的脱靶克隆池(最终形成由不需要的序列组成的克隆寡核苷酸池)的示例:

使用 Twist 产品,您再也不必担心嵌合了!查看以下示例,获得更直观的了解:
传统扩增与 Twist 内部扩增方法的性能对比

规格
| 寡核苷酸长度高达 300nt |
| 寡核苷酸池大小 池大小无限制 |
| 每个寡核苷酸平均产量 > 0.2 fmol |
| 均匀度 > 90% 寡核苷酸(在平均值的 < 5.0 倍范围内) |
| 嵌合率低至 1.5%* |
| 周转时间缩短至 4 周* |
*条款和条件:克隆寡核苷酸池(包括最多 100 个和 300 个核苷酸的克隆和扩增)的基线周转时间分别为 4-6 周和 6-8 周。对于所有需要新载体加入的克隆寡核苷酸池,无论长度如何,此基线周转时间将额外增加 2 周。嵌合率、丢失率和均一性将会根据序列复杂性而产生变化。
告诉我们您想详细了解哪些方面的信息。我们可以为您提供帮助!
联系我们
用于 CRISPR 筛选的向导 RNA 文库
CRISPR 筛选涉及基因活性的精确性、高通量敲除、干扰或调节,可揭示复杂的基因型-表型相互作用。寡核苷酸池旨在为每个基因编码多个向导 RNA 分子。
产品优势包括:
- 完全控制向导文库的设计,避免不必要的向导分子、测序量浪费和有限的向导/基因比率。
- 高保真 Cas 酶结合高精度合成,可实现真正的高保真筛选,且将脱靶效应降至最低。
- 极为均一、无偏差的池可放大信噪比,实现更多突变检测,从而降低了下游成本。
- 高达 300 nt 的寡核苷酸可兼容其他实验元素。根据寡核苷酸、基因组同源性或最少启动子,轻松添加多个向导。
观看我们的网络研讨会: 基于单细胞转录组学的新一代 CRISPR 筛选
基因组陈列式文库
陈列式文库可对基因组功能进行全面研究。寡核苷酸池可编码肽阵列、mRNA 编码序列或基因组片段。
产品优势包括:
- 精准陈列基因组的目标区域。在设计步骤中忽略不必要的序列,以此降低实验干扰。
- 通常无法合成为基因的复杂、重复或回文序列,可以在寡核苷酸中编码。
- 较大的肽结构或基因组结构可在长度为 300 nt 的寡核苷酸中编码。
- 均一的合成可大幅减少全面筛选所需的过采样。
阅读我们的文章: 使用 Twist 寡核苷酸池鉴定用于 RNA 疫苗开发的冠状病毒致病因子
高通量遗传控制筛选
了解控制基因表达和调控的遗传因子在合成生物学和潜在疾病疗法开发中的广泛应用。寡核苷酸池支持遗传控制的高通量筛选,包括基因表达谱分析和报告解读应用。
产品优势包括:
- 高质量的池确保您可以研究每个碱基的功能
- 高度均一性意味着您无需进行过多筛选就能找到答案
- 高达 300 nt 的池使您可以灵活地添加多个元素或同源区域
观看我们的网络研讨会: TechNetworks 上的表观转录组筛选
荧光原位杂交
荧光原位杂交(FISH)可以直接观察到生物样本中核酸的丰度和空间组织情况。寡核苷酸池可以编码与荧光标记结合的探针,从而对大量靶标进行可调整、可编程的可视化。
产品优势包括:
- 快速的 FISH 探针开发
- 序列控制可实现有效的靶向定位
- 更长的寡核苷酸可实现更多特异性靶向
观看我们的网络研讨会: 利用 Nicola Crosetto 方法处理的寡核苷酸池快速高效地生产 FISH 探针
用于 CRISPR 筛选的向导 RNA 文库
CRISPR 筛选涉及基因活性的精确性、高通量敲除、干扰或调节,可揭示复杂的基因型-表型相互作用。寡核苷酸池旨在为每个基因编码多个向导 RNA 分子。
产品优势包括:
- 完全控制向导文库的设计,避免不必要的向导分子、测序量浪费和有限的向导/基因比率。
- 高保真 Cas 酶结合高精度合成,可实现真正的高保真筛选,且将脱靶效应降至最低。
- 极为均一、无偏差的池可放大信噪比,实现更多突变检测,从而降低了下游成本。
- 高达 300 nt 的寡核苷酸可兼容其他实验元素。根据寡核苷酸、基因组同源性或最少启动子,轻松添加多个向导。
观看我们的网络研讨会: 基于单细胞转录组学的新一代 CRISPR 筛选
基因组陈列式文库
陈列式文库可对基因组功能进行全面研究。寡核苷酸池可编码肽阵列、mRNA 编码序列或基因组片段。
产品优势包括:
- 精准陈列基因组的目标区域。在设计步骤中忽略不必要的序列,以此降低实验干扰。
- 通常无法合成为基因的复杂、重复或回文序列,可以在寡核苷酸中编码。
- 较大的肽结构或基因组结构可在长度为 300 nt 的寡核苷酸中编码。
- 均一的合成可大幅减少全面筛选所需的过采样。
阅读我们的文章: 使用 Twist 寡核苷酸池鉴定用于 RNA 疫苗开发的冠状病毒致病因子
高通量遗传控制筛选
了解控制基因表达和调控的遗传因子在合成生物学和潜在疾病疗法开发中的广泛应用。寡核苷酸池支持遗传控制的高通量筛选,包括基因表达谱分析和报告解读应用。
产品优势包括:
- 高质量的池确保您可以研究每个碱基的功能
- 高度均一性意味着您无需进行过多筛选就能找到答案
- 高达 300 nt 的池使您可以灵活地添加多个元素或同源区域
观看我们的网络研讨会: TechNetworks 上的表观转录组筛选
荧光原位杂交
荧光原位杂交(FISH)可以直接观察到生物样本中核酸的丰度和空间组织情况。寡核苷酸池可以编码与荧光标记结合的探针,从而对大量靶标进行可调整、可编程的可视化。
产品优势包括:
- 快速的 FISH 探针开发
- 序列控制可实现有效的靶向定位
- 更长的寡核苷酸可实现更多特异性靶向
观看我们的网络研讨会: 利用 Nicola Crosetto 方法处理的寡核苷酸池快速高效地生产 FISH 探针